Saat ini, tubuh manusia dipandang tidak hanya sebagai organisme yang kompleks, tetapi juga sebagai lingkungan di mana mikroorganisme yang tak terhitung jumlahnya hidup. Jumlah gen mikroorganisme dalam tubuh manusia kira-kira 150 kali jumlah gen manusia. Mikroorganisme ini hidup dan berinteraksi dengan tubuh manusia dalam hubungan simbiosis yang berbeda, misalnya mutualisme, komensalisme atau bahkan parasitisme. Interaksi ini mungkin dikaitkan dengan gangguan fisiologis normal. Oleh karena itu, studi microbiome dapat memberikan pemahaman yang lebih baik tentang perkembangan penyakit manusia.
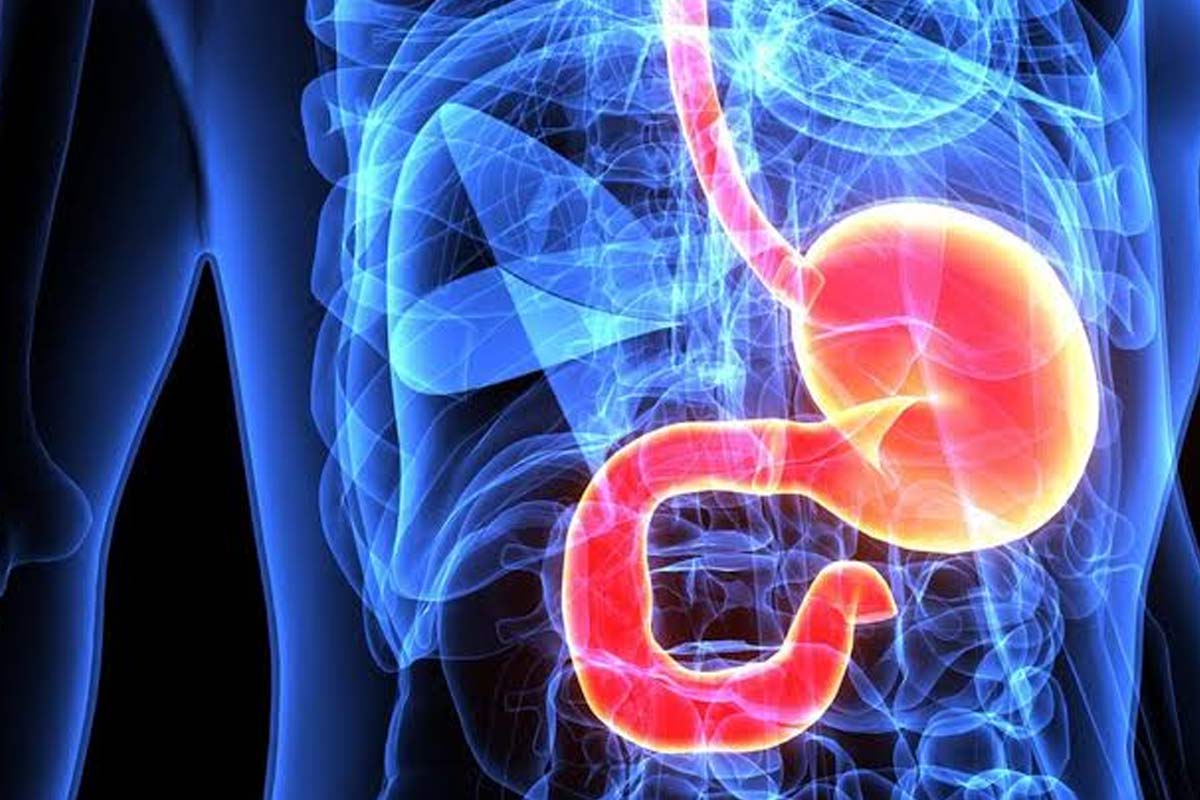
Perut manusia pernah dianggap sebagai lingkungan yang ‘steril’ karena keasamannya yang sangat tinggi. Hal ini kemudian dianggap sebagai penghalang gastrointestinal yang paling penting untuk berbagai mikroorganisme patogen. Namun, teori ini dibantah ketika Helicobacter pylori berhasil diisolasi dari perut manusia oleh Barry Marshal dan Robin Warren pada awal 1980-an. Terobosan ini menunjukkan bahwa mikroorganisme lain mungkin bisa bertahan dan menginfeksi perut manusia. Namun, upaya masa lalu untuk menggambarkan mikrobiota lambung terhambat oleh keterbatasan kultur konvensional, histologi dan imunohistokimia. Metode kultur membutuhkan upaya besar untuk mengisolasi dan mengidentifikasi bakteri dan tidak dapat mengidentifikasi sebagian besar dari mereka. Oleh karena itu, kemungkinan besar jumlah mikrobiota yang sebenarnya di dalam perut dianggap remeh.
Perkembangan teknologi sekuensing telah menjelaskan mikrobiota lambung. Penerapan next-generation sequencing (NGS) adalah lompatan besar yang memungkinkan pergeseran ke pendekatan budaya-independen dan berfokus pada analisis sekuens untuk mengidentifikasi mikroorganisme perut. Microbiome dan transkriptomee kemudian digunakan untuk mengidentifikasi mikrobiota dan mikroorganisme yang hidup di perut.
Jenis studi lain, studi metabolome, saat ini digunakan untuk mendukung studi microbiome dan transkriptome. Studi metabolome adalah analisis metabolit yang komprehensif, di mana molekul yang dilepaskan oleh organisme ke lingkungan diidentifikasi dan diukur. Metabolome dianggap sebagai indikator paling langsung terhadap perubahan lingkungan.
Berdasarkan dari gambaran di atas, peneliti dari Departemen Ilmu Penyakit Dalam, Fakultas Kedokteran, RSUD Dr. Soetomo, Universitas Airlangga berhasil mempublikasikan hasil penelitiannya di salah satu jurnal Internasional terkemuka, yaitu Expert Reviews in Molecular Medicine. Dalam penelitian ini, peneliti merangkum pembaruan terkini tentang studi mikrobiota, microbiome, transkriptome, dan metabolome perut manusia dan meninjau pentingnya mengintegrasikan beberapa kumpulan data untuk mendapatkan pemahaman yang lebih baik tentang perkembangan penyakit lambung.
Penelitian ini melaporkan bahwa microbiome, studi komunitas mikroba di lingkungan tertentu, telah berkembang secara signifikan sejak Proyek Microbiome pada Manusia dimulai. Microbiome telah dikaitkan dengan perubahan dalam relung lingkungan dan perkembangan berbagai penyakit. Pengembangan teknologi throughput tinggi seperti NGS juga memungkinkan dilakukannya studi transkriptome, yang memberikan data profil fungsional yang akurat. Studi metabolome, yang menganalisis metabolit yang ditemukan di lingkungan, adalah indikator kondisi lingkungan yang paling langsung. Meskipun setiap kumpulan data memberikan informasi berharganya sendiri, integrasi beberapa kumpulan data memberikan pemahaman yang lebih dalam tentang hubungan antara host, agen, dan lingkungan. Oleh karena itu, analisis jaringan menggunakan beberapa kumpulan data dapat memberikan pemahaman yang lebih jelas tentang patogenesis penyakit.
Berdasarkan penelitian ini dapat disimpulkan bahwa integrasi beberapa kumpulan data yang diperoleh dari studi microbiome, transkriptome, dan metabolome akan memberikan solusi untuk beberapa tantangan saat ini. Penerapan metode individual mungkin tidak cukup untuk menyediakan data yang akurat untuk mendukung hipotesis karena keterbatasannya. Integrasi studi yang berbeda akan memungkinkan para peneliti untuk membangun dan menguji model aktivitas mikroba dan interaksi antar mikroba atau inang mikroba. Ini akan memungkinkan pemahaman yang lebih baik tentang hubungan antara lingkungan dan komunitas mikroba.
Penulis: Muhammad Miftahussurur
Informasi detail dari penelitian ini dapat dilihat pada link artikel berikut: https://www.cambridge.org/core/journals/expert-reviews-in-molecular-medicine/article/abs/integrating-microbiome-transcriptome-and-metabolomee-data-to-investigate-gastric-disease-pathogenesis-a-concise-review/D6AA08AAD327CC5B5A26316E222197DE